|
Verbesserte Voraussagen des Krankheitsergebnisses anhand von Genexpressionsprofilen? | ||||||||||||||
|
Als prognostische Kriterien sind hauptsächlich Alter, Tumorgröße, Nodalstatus, histologischer Tumortyp, pathologischer Grad, Hormonrezeptorstatus, Überexpression von ERBB2 und p53-Status gebräuchlich. Die Erfahrung zeigt, dass diejenigen Patientinnen von einer adjuvanten Chemotherapie am meisten profitieren, deren kliniko-pathologischen Merkmale eine eher ungünstige Prognose ausweisen. Andererseits sind auf dieser Basis keine gesicherten Erkenntnisse darüber zu erlangen, welchen Frauen mit frühem Brustkrebs eine unnötige und oft sehr belastende Therapie definitiv erspart werden kann. Ließe sich auch nur ein kleiner Teil solcher Patientinnen identifizieren, bedeutete das bereits einen großen Schritt nach vorne.
In den letzten beiden Jahrzehnten haben neue Entwicklungen auf dem Gebiet der zellulären und molekularen Biologie von Brustkrebs
zu einem tieferen Verständnis der genomischen Grundlagen der Krankheit beigetragen. Insbesondere Veränderungen in transkriptionellen
Programmen, die für die enorme biologische Diversität von Tumoren verantwortlich sind, lassen sich heute mit Microarray-Techniken
analysieren. Deren Leistungsfähigkeit ermöglicht die Erfassung der transkriptionellen Aktivität tausender Gene in einem Arbeitsgang.
Der deutlichste Unterschied im Gesamtprofil der Genexpression wird zwischen ER-positiven und ER-negativen Tumoren beobachtet [1, 2]. Bei ersteren werden anhand der Genexpressionsprofile zwei Subtypen, luminal A und B, unterschieden. In ähnlicher Weise lassen sich ER-negative Tumoren in drei weitere molekulare Subtypen untergliedern: normaler brustähnlicher, ERBB2 -positiver und basalähnlicher Typ. Als basalähnliche Mammakarzinome werden Tumoren bezeichnet, deren neoplastische Zellen durchweg Gene exprimieren, wie das auch in basalen/myoepithelialen Brustzellen der Fall ist. Hierzu zählen hochmolekulare Zytokeratine (ZK; ZK5/6, 14, 17), Vimentin, p-Cadherin, B-Krystallin, Fascin sowie die Caveoline 1/2 sowie verschiedene myoepitheliale Marker. Der basalähnliche Phänotyp war im Grunde bereits vor der molekulargenetischen Ära anhand des immunhistochemischen Nachweises von Zytokeratinen in Brusttumoren erkannt worden [4], geriet jedoch wieder weitgehend in Vergessenheit. Zu einem sehr hohen Prozentsatz werden in solchen Tumoren verschiedene mit der Proliferation im Zusammenhang stehende Gene überreichlich exprimiert. In bis zu 85 % der basalähnlichen Karzinome lässt sich entweder p53 immunhistochemisch nachweisen oder es finden sich p53-Genmutationen.
Basalähnliche Mammakarzinome weisen in ihren klinischen und Tumorcharakteristika große Übereinstimmung mit dreifach
rezeptornegativen Brusttumoren auf. Es handelt sich überwiegend um aggressive Tumoren. Die weitgehende Übereinstimmung
hat dazu geführt, dass beide Begriffe vielfach synonym verwendet werden.
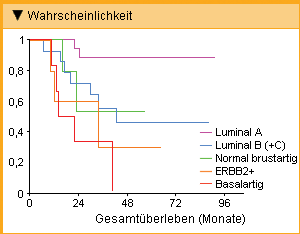
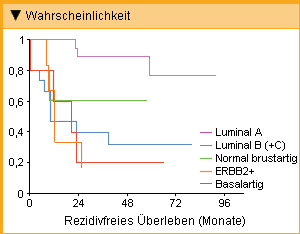
Im ersten Arbeitsgang wurden jeweils über 8 000 Gene analysiert. Die Klassifizierung von Subtypen erfolgte dann anhand einer Untergruppe von 496 Genen, bei denen die Expression zwischen verschiedenen Tumoren deutlich stärker variierte als zwischen paarweisen Proben eines Primärtumors oder eines Tumors und seinen Metastasen. Hierzu wurden hierarchische Clusteranalysen ohne Einbeziehung von Informationen über Krankheitsergebnisse durchgeführt. Die ursprüngliche Einteilung sah vier Subtypen vor – basalähnlich, ER+/luminal epithelial, ERBB2-überexprimierend und normal brustartig [2]. Die paarweisen Analysen von Primärtumor und Metastasen, wie auch die vom Primärtumor vor und nach einer Chemotherapie zeigten jeweils ähnliche Genexpressionsmuster. Daraus kann geschlossen werden, dass sich die molekulargenetische Programmierung des Primärtumors bei der Metastasierung nicht ändert [2]. Sorlie et al. (2001) verfeinerten die Klassifizierung in vier Subtypen, nachdem sie Proben von 78 Brusttumoren, drei Fibroadenomen und vier normalen Brustgeweben durch hierarchisches Clustern analysiert hatten. Ihre molekularen Profile lieferten insbesondere im Bereich der luminalen ER-positiven Tumoren über die in den klinisch determinierten Untergruppen hinausgehende Informationen. Hierbei wird deutlichen prognostischen Unterschieden durch die Unterteilung in luminal A und luminal B Rechnung getragen. Vom Typ luminal B ließ sich eine mögliche weitere Untergruppe, luminal C, abgrenzen. In der Studie von Sorlie et al. (2001) wurde ferner getestet, inwieweit sich die mittels hierarchischer Cluster identifizierten fünf Brustkrebs-Subtypen auch klinisch anhand des krankheitsfreien und Gesamtüberlebens unterscheiden. Hierbei war das Krankheitsergebnis bei Patientinnen mit Luminal-A-Tumoren sowohl hinsichtlich des Gesamtüberlebens (Abb 1), als auch hinsichtlich des rezidivfreien Überlebens (Abb. 2) deutlich günstiger als bei den anderen Brustkrebs-Subtypen [3].
Die Zahl der Gene, die signifikant im Zusammenhang mit dem Krankheitsergebnis standen, ließ sich auf 70 reduzieren. Mit dieser 70-Gen-Signatur konnten Patientinnen identifiziert werden, die eine adjuvante Chemotherapie benötigten, und solche, bei denen keine adjuvante Chemotherapie erforderlich war [4]. Die ungünstige Prognose-Signatur bestand aus überexprimierten Genen mit Funktionen bei der Zellzyklusregulierung, der Invasion, der Metastasierung und der Angiogenese. In derselben Arbeitsgruppe wurde die 70-Gen-Signatur anschließend auf ein Kollektiv von 295 Brustkrebs-Patientinnen angewandt. Diese Frauen waren jünger als 53 Jahre und hatten Brustkrebs der Stadien I oder II. Von ihnen waren 151 nodalnegativ und 144 nodalpositiv. Dabei konnte eine verbesserte Vorhersagbarkeit des Krankheitsergebnisses gegen-über der Prognose anhand von klinischen und histologischen Ergebnissen erzielt werden [5]. Buyse et al. (2006) überprüften anhand eines unabhängigen Patientinnen-Kollektivs, inwieweit die 70-Gen-Signatur zusätzlich zu kliniko-pathologischen Kriterien prognostische Informationen zu liefern imstande ist. Sie verwandten dazu Daten aus fünf europäischen Zentren. Die insgesamt 307 Patientinnen, bei denen während einer mittleren Nachbeobachtungszeit von 13,6 Jahren insgesamt 137 Ereignisse eingetreten waren, wurden sowohl anhand der Gen-Signatur als auch nach klinischen Risikomerkmalen jeweils als mit hohem oder niedrigem Risiko behaftet klassifiziert. In den Analysen erwies sich die 70-Gen-Signatur in ihren Voraussagen sowohl hinsichtlich der Zeitdauer bis zum Auftreten von Fernmetastasen als auch hinsichtlich des Gesamtüberlebens als deutlich überlegen (Hazard Ratios 2,32 bzw. 2,79). Die Untersucher kamen zu dem Ergebnis, dass die 70-Gen-Signatur bei frühem Brustkrebs unabhängige diagnostische Informationen liefert, die über die der kliniko-pathologische Risikobewertung hinausgehen [6].
Basierend auf der 70-Gen-Signatur wurde MammaPrint® als molekulardiagnostischer Test entwickelt. Hiermit soll das Risiko der
Metastasierung eines T1- oder T2-Mammatumors bei bis zu 55-jährigen Patientinnen bewertet werden. Allerdings muss sich die Anwendbarkeit
des Tests in der klinischen Praxis zunächst in prospektiven klinischen Prüfungen erweisen. Diesbezüglich wird in Europa gegenwärtig MINDACT
(Microarray In Node-negative and 1 to 3 positive lymph node Disease may Avoid ChemoTherapy) als prospektive randomisierte Studie zum Vergleich
der 70-Gen-Signatur mit herkömmlichen kliniko-pathologischen Kriterien gestartet.
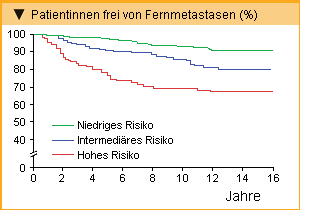
Die Analyse der Tumorproben von 668 Patientinnen ergab in 55 % der Fälle ein niedriges (Rezidiv Score <18), in 24 % der Fälle ein intermediäres (Rezidiv Score 18-30) und in 27 % der Fälle ein hohes Rezidivrisiko (Rezidiv Score >30). In der Gruppe mit niedrigem Rezidivrisiko waren nach zehn Jahren noch 93,4 % der Patientinnen ohne Fernrezidiv, während das in der Gruppe mit hohem Rezidivrisiko nur bei 69,5 % der Frauen der Fall war (p<0,001; Abb. 3). Der Rezidiv Score stand auch signifikant im Zusammenhang mit dem Rezidiv-freien Intervall und dem Gesamtüberleben.
Vom 21-Gen Assay gibt es mit dem OncotypeDX™ eine kommerzielle Form, die vom Clinical Laboratory Improvement Amendments (CLIA) für Patientinnen mit nodalnegativem, ER-positiven Brustkrebs nach Tamoxifentherapie zugelassen ist. Im Gegensatz zum MammaPrint® kann beim OncotypeDX™ auch Formalin-fixiertes, archiviertes, in Paraffin eingebettetes Tumorgewebe verwendet werden. Zur Validierung des OncotypeDX™ wurde die Trial Assigning IndividuaLized Options for Treatment (Rx) bzw. TAILORx (Versuch zur Bestimmung von individualisierten Therapiemöglichkeiten), bereits im Mai 2006 gestartet. Dennoch sind Ergebnisse nicht vor 2010 zu erwarten. Demzufolge wurde die Frage, ob die molekularen Assays (MammaPrint®, OncotypeDX™) die Entscheidung für oder wider eine adjuvante Chemotherapie bei Frauen mit einem frühem Mammakarzinom beim 11. Internationalen Brustkrebskongress in St. Gallen 2009 kontrovers diskutiert.
[1] Stadler ZK, Come SE, 2009. Review of gene-expression profiling and its clinical use in breast cancer. Crit Rev Oncol Hematol 69:1-11. [2] Perou CM, Sorlie T, Eisen MB, et al. 2000. Molecular portrait of human breast tumours. Nature 406:747-752. [3] Sorlie T, Perou CM, Tibshirani R, et al. 2001. Gene expression patterns of breast carcinomas distinguish tumor subclasses with clinical implications. Proc Natl Acad Sci USA 98:10869-10874. [4] van ´t Veer LJ, Dal H, van de Vijver MJ, et al. 2002. Gene expression profiling predicts clinical outcome of breast cancer. Nature 415:530-536. [5] Buyse M, Loi S, Van´t Veer LJ, et al. 2006. Validation and clinical utility of a 70-gene prognostic signature for women with node-negative breast cancer. J Natl Cancer Inst 98:1183-1192. [6] van de Vijver MJ,He YD, van´t Veer LJ, et al. 2002. A gene-expression signature as a predictor of survival in breast cancer. N Engl J Med 347:1999-2009. [7] Paik S, Shak S, Tang G, et al. 2004. A multigene assay to predict recurrence of tamoxifen-treated, node-negative breast cancer. N Engl J Med 351:2817-2826. [8] Sotiriou C, Wirapati P, Loi S, et al. 2006. Gene expression profiling in breast cancer: understanding the molecular basis of histologic grade to improve prognosis. J Natl Cancer Inst 98:262-272. | ||||||||||||||
|
Früher Brustkrebs: Verbesserte Voraussagen des Krankheitsergebnisses anhand von Genexpressionsprofilen? Schlüsselwörter: Früher Brustkrebs, Genexpressionsprofile, 70-Gen-Signatur, MammaPrint, 21-Gen Score, OncotypeDX, MINDACT |
||||||||||||||
| ||||||||||||||